CRISPR-Cas技术促进了生物医学研究。除了广泛使用的Cas9系统之外1,其他CRISPR亚型也不断被发现并应用于基因编辑,例如能够装载进AAV病毒的SaCas9(1053 aa)2以及更小的微型CRISPR-Cas系统Cas12f(400~550 aa)3-6。已发表的工作重建了CRISPR-Cas系统的起源,发现原核转座子编码的IscB和TnpB蛋白分别是Cas9与Cas12核酸酶的祖先7。这些祖先蛋白尺寸较小,但其是否具备核酸酶活性缺乏证据;直到2021年,IscB和TnpB被发现在非编码RNA(omegaRNA或reRNA)引导下切割双链DNA,证实了其与CRISPR-Cas系统相似的工作机制8, 9。TnpB由IS200/IS605等原核转座子家族编码,并被推测参与转座子的扩张9。TnpB的分布非常广泛,在目前已知的基因组存在超过百万的拷贝8;但此前的研究只发现了一种在人类细胞中具有活性的TnpB核酸酶(ISDra2),且效率不高9;因此,TnpB这一有潜力作为微型编辑工具的多样性宝库急需系统性的挖掘和研究。同时,由于可能推动转座子扩张,TnpB靶向切割DNA所依赖的关键元件(如reRNA)与转座子或存在关联,因而可以基于转座子信息进行预测,这将为工具的开发提供便利。
2023年6月29日,中国科学院动物研究所/北京干细胞与再生医学研究院王皓毅研究员、项光海博士和中国科学院动物研究所张勇研究员团队合作在Nature Biotechnology杂志在线发表题为Evolutionary mining and functional characterization of TnpB nucleases identify efficient miniature genome editors的研究论文。Nature Biotechnology杂志同时发表了Research Briefing文章对该研究成果进行了总结和展望(Hypercompact genome editors are discovered by mining a transposon family)。该研究创新性地建立了对TnpB相关靶向基因编辑系统的大规模挖掘方法,并首次对多样性极其丰富的TnpB核酸酶进行了大规模挖掘,从而鉴定到了33个在原核系统具有靶向编辑活性的TnpB蛋白,其中5个在真核系统具有活性。
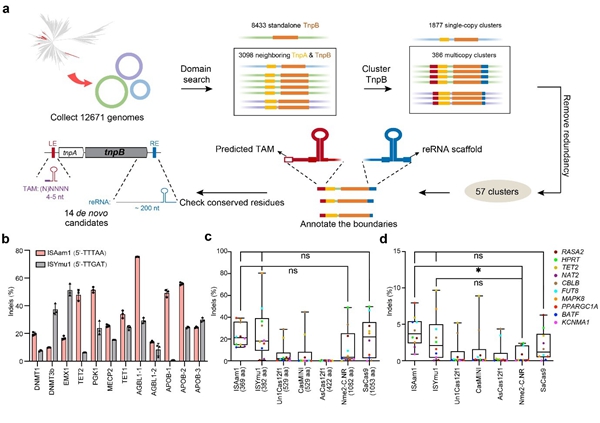
研究团队首先对ISfinder原核转座子数据库中IS605编码的TnpB蛋白进行了全面的分析和挖掘,从64个候选项中鉴定出了25种在大肠杆菌中活跃的系统,其中3种在人类细胞中具有基因编辑活性。对于功能数据的进一步分析则揭示TnpB蛋白相关的reRNA骨架与IS200/IS605转座子的RE序列具有完全重叠的3’末端;而TAM序列则与转座子上游的插入位点序列相同。该结果表明在TnpB系统中,与RNA介导的编辑器相关的三大要素(核酸酶、gRNA骨架和TAM序列)均可通过生物信息分析准确预测;这为大规模筛选高活性TnpB核酸酶奠定了基础。这一发现同时也进一步确定了TnpB在IS605中的功能,即作为归巢核酸酶切割转座之后的原位点,从而诱导重组修复实现转座子的拷贝数扩增。
研究团队随后从4个方面对TnpB相关的reRNA骨架进行了分析,结果表明:1)reRNA骨架在120-300nt的长度范围内均能够有效发挥功能,而120-140nt的reRNA骨架活性最强;2)reRNA骨架在3’末端的碱基对其功能有重要影响,单一碱基的突变即会显著降低编辑活性;3)靶向序列的长度在16-20nt为最佳;4)靠近TAM端的12nt是TnpB编辑器的核心序列。研究者进一步整合分析了影响TnpB编辑器活性的潜在因素,发现来自细菌的、由多拷贝转座子编码的、具有完整蛋白结构域和保守氨基酸的TnpB编辑器更为活跃。
最后,团队基于上述理解,建立了大规模挖掘全新TnpB基因编辑器的方法(下图),对部分未经转座子注释的原核基因组进行了从头注释和功能预测,并直接在人类细胞系中筛选获得了新的微型高活性TnpB编辑器ISAam1(369 aa)和ISYmu1(382 aa)。和其他微型Cas蛋白的平行比较发现,ISAam1和ISYmu1的活性与SaCas9相当,显著高于数种已报道的Cas12f蛋白及其变体。
综上,该研究建立了适用于TnpB编辑器的大规模筛选体系,进一步证明了TnpB在转座子扩张中的功能,并对这一类编辑器进行了系统的功能解析,从而获得了目前最小的具备原创知识产权的微型基因编辑器。考虑到体内基因治疗和细胞治疗经常因Cas蛋白过大而递送受限,这一成果将推动相关方面的研究和临床应用。
该研究由中国科学院动物研究所/北京干细胞与再生医学研究院完成。北京干细胞与再生医学研究院/中国科学院动物研究所王皓毅研究员、项光海博士和中国科学动物研究所张勇研究员为文章的共同通讯作者。项光海博士、研究生李源清、孙晶、霍雍元为文章的共同第一作者。王皓毅研究员长期致力于新型基因编辑工具的开发(Cell Discovery 2019)及CAR-T细胞治疗研究(Cancer Cell 2022);张勇研究员则致力于转座子等机制介导的新重复基因的起源和进化研究(Nature Ecology & Evolution 2022、Nature Communications 2021)。两个团队的合作推动了对TnpB的挖掘。该研究得到了科技部、中国科学院先导专项、农业农村部和国家自然科学基金委等项目的资助。
参考文献:
1. Hsu, P.D., Lander, E.S. & Zhang, F. Development and applications of CRISPR-Cas9 for genome engineering. Cell 157, 1262-1278 (2014).
2. Ran, F.A. et al. In vivo genome editing using Staphylococcus aureus Cas9. Nature 520, 186-191 (2015).
3. Bigelyte, G. et al. Miniature type V-F CRISPR-Cas nucleases enable targeted DNA modification in cells. Nat Commun 12, 6191 (2021).
4. Xu, X. et al. Engineered miniature CRISPR-Cas system for mammalian genome regulation and editing. Mol Cell 81, 4333-4345 e4334 (2021).
5. Wu, Z. et al. Programmed genome editing by a miniature CRISPR-Cas12f nuclease. Nat Chem Biol 17, 1132-1138 (2021).
6. Kim, D.Y. et al. Efficient CRISPR editing with a hypercompact Cas12f1 and engineered guide RNAs delivered by adeno-associated virus. Nat Biotechnol 40, 94-102 (2022).
7. Kapitonov, V.V., Makarova, K.S. & Koonin, E.V. ISC, a Novel Group of Bacterial and Archaeal DNA Transposons That Encode Cas9 Homologs. J Bacteriol 198, 797-807 (2015).
8. Altae-Tran, H. et al. The widespread IS200/IS605 transposon family encodes diverse programmable RNA-guided endonucleases. Science 374, 57-65 (2021).
9. Karvelis, T. et al. Transposon-associated TnpB is a programmable RNA-guided DNA endonuclease. Nature 599, 692-696 (2021).